Dra. Bertha E. Cocotle Ronzón. José Arenas Benhumea.
Departamento de Patología Experimental Fac. Medicina U.V.
Desde que Watson y Crick publicaron el modelo
de la estructura en doble hélice del ácido desoxirribonucleico o
ADN, se ha producido una verdadera revolución que ha llevado
a crear una nueva rama de la ciencia, la Biología Molecular,
cuyo objetivo fundamental es la comprensión de todos aquellos
procesos celulares que contribuyen a que la información
genética se transmita eficientemente de unos seres a otros
y se exprese en los nuevos individuos, es decir, aquellos procesos
celulares en los que participan los ácidos nucleicos;
por tanto utiliza varias herramientas y técnicas para
su estudio.
Los ácidos nucleicos son
el ácido
desoxirribonucleico (ADN) y el ácido ribonucleico (ARN),
se consideran como polímeros de alto peso molecular, y
se localizan principalmente en el núcleo de las células,.
Están constituidos
por nucleótidos formados por una molécula de azúcar
(ribosa para el ARN y desoxirribosa para ADN), una base orgánica
nitrogenada (adenina, guanina, citosina y timina para el ADN
y adenina, guanina, citosina y uracilo para el ARN), y un grupo
fosfato.
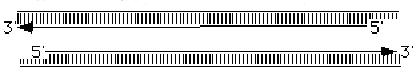
El ADN es una estructura de forma
helicoidal constituida por dos cadenas complementarias entre si;
en la parte
externa
se
localizan las moléculas de azúcar-fosfato y en
la parte central se encuentran las bases orgánicas nitrogenadas
unidas las de una cadena con las de la otra, a través
de puentes de hidrógeno, lo que da así estabilidad
a la doble hélice; las dos cadenas tienen sentidos opuestos,
una va en sentido 5’ ? 3’ y la otra lo hace en sentido
3’ ? 5’.
El ADN regula la naturaleza y composición
de las células,
transmite la información hereditaria, determina la estructura
de las proteínas y a través de enzimas controla
el resto de las funciones celulares.
Estas actividades las lleva a cabo
al realizarse una copia de un fragmento de la molécula
o de toda, ya sea que se requiera la síntesis de una proteína
o la transmisión
de la información genética de la célula.
Para que ambas situaciones se lleven a cabo eficientemente, las
dos cadenas de ADN deben separarse.

Todas las enfermedades genéticas y algunas no heredadas
se asocian a cambios en la molécula de ADN; estos padecimientos
se pueden analizar en términos moleculares, con lo que,
eventualmente, algunos podrán ser tratados o curados al
detectarse el defecto que exista.
La reacción en cadena de
polimerasa o PCR (de sus siglas en inglés Polymerase Chain
Reaction) fue descubierta en 1985 por Karry Mullis en el laboratorio
de
Cetus
Corporation,
California, USA, lo que le representó el premio Nobel
en 1993. Su nombre lo debe a que la actividad de la enzima ADN
polimerasa permite fabricar una cadena de ADN complementaria
a otra ya existente. Es una técnica empleada en Biología
Molecular que permite la copia in vitro de fragmentos específicos
de ADN; consiste en la separación por calor de las dos
cadenas del ADN que se quiere amplificar a partir de un punto
marcado por dos segmentos pequeños de ADN conocidos como
cebadores (primers o iniciadores), que están constituidos
de 10 a 30 nucleótidos e indican el inicio y el final
del fragmento a duplicar. Mediante la acción de una enzima
denominada ADN polimerasa, se van agregando en forma complementaria
los nucleótidos necesarios para obtener una replica exacta
de la cadena original. Este procedimiento se repite varias veces
y se obtienen múltiples copias del fragmento de ADN escogido.
Sus únicos requerimientos
son que existan nucleótidos
en el medio (adenina, guanina, citosina y timina), cebadores,
ADN original y la enzima DNA polimerasa. Debido a que para el
desarrollo de la técnica se emplean temperaturas mayores
de 70°, se requiere de una enzima que no se inactive a temperaturas
elevadas, por lo que se emplea la DNA polimerasa de la bacteria
thermus aquaticus que vive en aguas termales y cuya enzima puede
trabajar a altas temperaturas.
La reacción en cadena de
polimerasa se desarrolla en tres etapas:
1ª. – Desnaturalización
del ADN. Por medio de la aplicación de calor a 94°C, se produce la separación de las dos cadenas de la molécula de DNA que se quiere amplificar. Al romperse los enlaces de hidrógeno, cada cadena actúa como molde para fabricar su complementaria. |
|
Paso
1: Desnaturalización
del ADN. (95oC-98oC) |
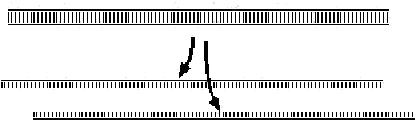 |
2ª. – Hibridación
con cebadores. La temperatura se disminuye rápidamente a 55° C, para lograr que los cebadores o primers “reconozcan” sus secuencias complementarias en las cadenas de DNA correspondientes y se unan con ellas. Los primers no deben ser complementarios entre sí y deben corresponder a los extremos del fragmento del DNA que se quiere amplificar. |
|
Paso
2: Hibridación
con iniciadores (45oC-65oC) |
 |
3ª. – Extensión
con Taq polimerasa.
Para esta etapa, la temperatura se eleva a 72° C, con lo que la taq polimerasa agrega los diferentes nucleótidos complementarios siguiendo el orden de la cadena que sirve de molde. |
|
Paso
3: Extensión
con Taq polimerasa
(65oC-75oC) |
 |
Después
de completar el ciclo 2 tendremos cuatro copias.
|
 |
El proceso se repite
un número determinado
de veces o ciclos, generalmente 30, y se consigue un aumento
o amplificación exponencial del número de copias
del fragmento de ADN que sirvió como molde. (1, 2, 3)
Una ventaja de la técnica
es que amplifica únicamente
el fragmento
de ADN que queremos, aunque esté en cantidades mínimas (alta sensibilidad)
o en presencia de grandes cantidades de ADN semejantes (alta especificidad).
La reacción es eficaz incluso si se parte de muestras de ADN muy poco
purificadas, es decir, que se encuentran en presencia de otros componentes.
Una vez amplificada la molécula del DNA que se
desea, se coloca la muestra en gel de poliacrilamida, en el que se agrega azul
de bromofenol que nos permitirá visualizar
cómo la muestra corre a través del gel, para lo que se emplea corriente
eléctrica a 100 mV, durante 30 a 40 minutos.
De acuerdo con el tamaño de la molécula de ADN, será la
distancia que correrá en el gel; posteriormente este se tiñe con
bromuro de etidio que permite visualizar la extensión en forma de bandas
con el empleo de luz ultravioleta. La banda obtenida se compara con un marcador
de peso molecular para poder establecer su tamaño y así verificar
si corresponde al fragmento que se busca o se puede correr al mismo tiempo que
un control conocido y establecer comparaciones entre ambas bandas. (fig 1).
El material biológico a partir del cual se pueden obtener muestras para
el análisis del DNA es múltiple e incluye sangre, piel, semen,
cabellos, material de biopsia, o puede conseguirse a partir de los instrumentos
utilizados para la recolección de las muestras como son cotonetes o cepillos.
La PCR se aplicó inicialmente como una técnica de laboratorio de
investigación, pero su gran especificidad y sensibilidad ha permitido
el desarrollo de técnicas específicas basadas en ella con aplicación
en muchos campos de la investigación y el análisis. Así,
se ha convertido en una herramienta fundamental en campos tan diversos como la
investigación (clonación de genes, detección de clones recombinantes,
estudio de ADN de fósiles), medicina forense y criminalística (determinación
de paternidad, identificación de individuos y de sospechosos, así como
en el análisis de la evidencia en casos de asesinato, violación,
etc.), sanidad animal y mejora genética (detección de enfermedades
genéticas e infecciosas, pureza de razas, etc.) y medicina (diagnóstico
de enfermedades ).
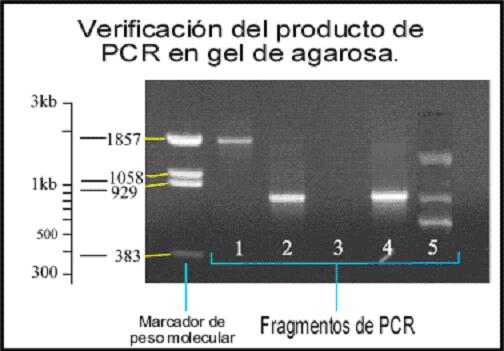
Figura 1. Migración en gel de los pares de bases de una molécula de DNA amplificada.
En esta última, la PCR se encuentra en
un amplio campo de investigación y desarrollo, por lo
que se ha empleado en la exploración de enfermedades genéticas
como en casos de anemia de células falciformes, fibrosis
quística, distrofia miotónica y el Síndrome
del cromosoma X frágil. (4) Además se ha utilizado
para el diagnóstico prenatal, creación de mapas
genéticos y en la detección de esperma aneuploide.
(5)
También se ha utilizado
para realizar la subtipificación
de neoplasias, para vigilar la respuesta al tratamiento y en
la detección de las recurrencias o de recaídas
tempranas (se menciona que tiene una sensibilidad para detectar
una sola célula tumoral entre un millón de células
sanas), lo que permite establecer un pronóstico de la
enfermedad. (6)
Dentro de este campo, también se ha utilizado para el
estudio de la estructura y expresión de los oncogenes,
y para detectar el reordenamiento de tumores humanos con anomalías
del cariotipo, así como de anomalías de la expresión
genética en ausencia de alteraciones del cariotipo. Una
de las aplicaciones más útiles de la PCR en la
oncología pediátrica es en la detección
de traslocaciones cromosómicas. (7,8)
En el campo del laboratorio clínico y de aplicación
en la endocrinología, se ha utilizado para detectar la
producción de hormonas ectópicas. (9)
Dentro de la microbiología e infectología, se ha
utilizado para identificar el DNA de parásitos, virus
o bacterias, incluyendo también la detección de
resistencia a los antibióticos. (10) De la misma forma
puede detectar rápida y exactamente la presencia de agentes
infecciosos de lento crecimiento como Chlamydia trachomatis,
Toxoplasma gondii, Pneumocystis carinii, virus de la inmunodeficiencia
humana, Mycobacterium tuberculosis, Micoplasmas, herpesvirus,
citomegalovirus, virus de la Hepatitis C, por lo que se puede
emplear en: diagnóstico rápido de infecciones víricas,
detección de microorganismos en células infectadas
o transformadas, determinación de la relación genética
entre varios tipos de microorganismos similares, para correlacionar
la presencia de agentes virales o infecciosos en tejidos neoplásicos,
determinar la resistencia a los antibióticos de las bacterias,
además de detectar y cuantificar microorganismos difíciles
de cultivar o para los que no existen reactivos serológicos.
La técnica de PCR tiene el potencial de realizar una rápida
detección del DNA bacteriano en muestras de sangre, y
evita los contratiempos encontrados con los cultivos sanguíneos
en el diagnóstico de bacteremia (11).
Otro aspecto importante de la prueba es que se ha utilizado para
determinar si los individuos son genéticamente compatibles
en caso de transplante renal, con lo que se disminuye de forma
importante la posibilidad de rechazo.
Conclusiones
La técnica de la PCR presenta ventajas sobre las técnicas
convencionales de diagnóstico debido a que tiene una mayor
sensibilidad y especificidad, presenta un menor número
de falsos positivos y negativos, emplea un tiempo mucho menor
para obtener los resultados y puede utilizarse en todas las
ramas de la medicina.
Por lo tanto, los médicos deben familiarizarse con esta
técnica, su empleo e interpretación, para poder
proporcionarle beneficios a los pacientes.
Bibliografía
-
Lodish H, Baltimore D. “Recombinant DNA technology”. En: Lodish H, Baltimore D. Molecular Cell Biology. Scientific American books. 1997: 254-6.
-
Larrick JW. The PCR Technique: Quantitative PCR. Biotechniques Books. Eaton Publishing. 1997
-
Siebert P. The PCR Technique. RT-PCR. Biotechniques Books. Eaton Publishing. 1997
-
Thornton C. “The myotonic dystrophies”. Semin Neurol 1999; 19: 25 – 33
-
Thornhill, AR Snow K. 2Molecular Diagnostics in Preimplantation Genetic Diagnosis”. J Mol Diagn 2002 4: 11-29.
-
Lindblom A, Liljegren A. Tumor markers in malignancies. BMJ 2000; 320: 424 – 7.
-
Rowland, JM. “Molecular genetic diagnosis of pediatric cancer: current and emerging methods”. Pediatric Clinics of North America. 2002; 49 (6): 1415.
-
Hirose Y, Aldape K, Takahashi M, Berger MS, and Burt GF. “Tissue Microdissection and Degenerate Oligonucleotide Primed-Polymerase Chain Reaction Is an Effective Method to Analyze Genetic Aberrations in Invasive Tumors”. J Mol Diagn 2001 3: 62-67.
-
White PC, Speiser PW: “Congenital adrenal hyperplasia due to 21-hydroxylase deficiency”. Endocr Rev 2000, 21:245-291
-
Louie M, Louie L, Simor A. “The role of DNA amplification technology in the diagnosis of infectious diseases”. CMAJ 2000; 163 (3): 301 – 9.
-
Teba L. “Polymerase chain reaction: A new chapter in critical care diagnosis”. Critical Care Medicine 1999; 27:860-86.

